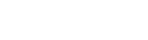16. Dez 2019
Lesedauer 15 Min.
Herzsimulator 1.0
Forschung
Von wandernden Ionen über das Zellgewebe bis hin zur Aufzeichnung eines EKG: Die Softwaresimulation des menschlichen Herzens ermöglicht maßgeschneiderte Therapien.
Am Karlsruher Institut für Technologie (KIT) haben Forscher ein Computermodell des menschlichen Herzens entwickelt. Die Wissenschaftler sind mittlerweile sogar schon so weit, dass sie das Modell auf die individuellen Eigenschaften eines einzelnen Patienten maßschneidern können. Diese Simulation hat einen erheblichen Nutzen für die medizinische Praxis.
Blick über den Tellerrand
Die Idee zu diesem Artikel ist: Mal raus aus dem Gewohnten. Den Blick über den Tellerrand in die Forschung wagen. Ein solcher Ausflug in die Forschung will keine Lösungen für alltägliche Programmierprobleme bieten, sondern die Gedanken anregen. Vielleicht bleibt dabei die eine oder andere Idee für die eigene Arbeit hängen. Im besten Fall erweitert es den Horizont und stimuliert die eigene Kreativität.
An einem solchen maßgeschneiderten Computermodell kann ein Arzt beispielsweise Experimente durchführen, ohne dadurch die Gesundheit eines Patienten zu gefährden. Bevor ein Arzt also zum Beispiel einen chirurgischen Eingriff am Herzen vornimmt oder eine medikamentöse Behandlung verschreibt, kann er die Auswirkungen der geplanten Therapie zunächst in einem individuell angepassten Computermodell simulieren.Auf diese Weise kann der Arzt bereits im Voraus erkennen, ob die geplante Behandlung möglicherweise unerwünschte Nebenwirkungen verursachen würde. Wenn dem so ist, kann er die geplante Therapie so oft modifizieren und in der Simulation wiederholen, bis der gewünschte Therapieeffekt eintritt. Erst dann nimmt der Arzt die Behandlung auch am realen Patienten vor.Das hat die dotnetpro interessiert. Wir haben uns gefragt: Wie ist es gelungen, ein Modell des menschlichen Herzens so realitätsnah zu entwickeln?
Fantasie …
Unwillkürlich fängt man wahrscheinlich zunächst an, in Gedanken selbst eine solche Simulation zu entwickeln.Letztlich sind für jede Computersimulation immer zwei Schritte erforderlich:- Zunächst braucht man von dem realen Gegenstand ein abstraktes Modell.
- Anschließend muss das abstrakte Modell in Code umgesetzt werden.
Kleines Herz-Glossar
<b>Ablation </b>
Wenn man das Modell so aufbauen könnte, aus einzelnen Zellen, die miteinander in Wechselwirkung stehen, dann wäre es eine ideale Vorlage für die objektorientierte Programmierung. Es gibt eine Klasse für eine Zelle. Diese Klasse hat bestimmte Eigenschaften und spezielle Methoden für die Interaktion mit der Umgebung.Für ein ganzes Herz wird man dann einige Millionen oder Milliarden solcher Zellen benötigen, also für jede simulierte Zelle eine Instanz der entsprechenden Klasse. Hier könnte die objektorientierte Programmierung mal so richtig zeigen, was sie kann!Aber das sind nur Tagtraumfantasien. Ein Besuch am Karlsruher Institut für Technologie soll zeigen, wie die Simulation tatsächlich gelingt.
… und Realität
Am Karlsruher Institut für Technologie finden die Forschungsarbeiten zur Herzsimulation am Institut für Biomedizinische Technik statt [1]. Dort ist Dr. Axel Loewe Leiter der Arbeitsgruppe Herzmodellierung. dotnetpro hat ihn getroffen und sich die Arbeiten am Institut erklären lassen.Im Interview, siehe Kasten Mit der Forschung im Gespräch, gibt er einen kleinen Einblick in die Arbeit seines Teams. Die Modellierung und Umsetzung des Systems soll im Folgenden beschrieben werden.„Im besten Fall haben wir sogar die Möglichkeit, die Therapie zu personalisieren.“
„Im besten Fall haben wir sogar die Möglichkeit, die Therapie zu personalisieren.“
Aufbau der Modellierung
Ganz am Anfang steht die Frage, welche Eigenschaften des Herzens überhaupt zu messen und anschließend zu simulieren sind, um zu einer aussagekräftigen Modellierung gelangen zu können.Die Karlsruher Forscher unterscheiden hier vier Ebenen:- Elektrophysiologie
- Elastomechanik
- Strömungsvorgänge
- Blutkreislauf
Elektrophysiologie
Die wichtigste Rolle bei der Modellbildung haben die elektrischen Vorgänge im Herzen. Die Karlsruher Forscher haben diesen Bereich seinerseits in vier Stufen aufgeteilt, die aufeinander aufbauen:- Die unterste Ebene bildet die Vorgänge auf molekularer Ebene ab. Herzzellen tauschen Ionen aus.
- Darüber stehen die Vorgänge innerhalb einer Zelle.
- Zahlreiche Zellen werden zum Gewebe des Herzens zusammengesetzt.
- Das Organ wird in den Körper eingefügt. Auf dieser Ebene lassen sich EKG-Ströme ableiten.
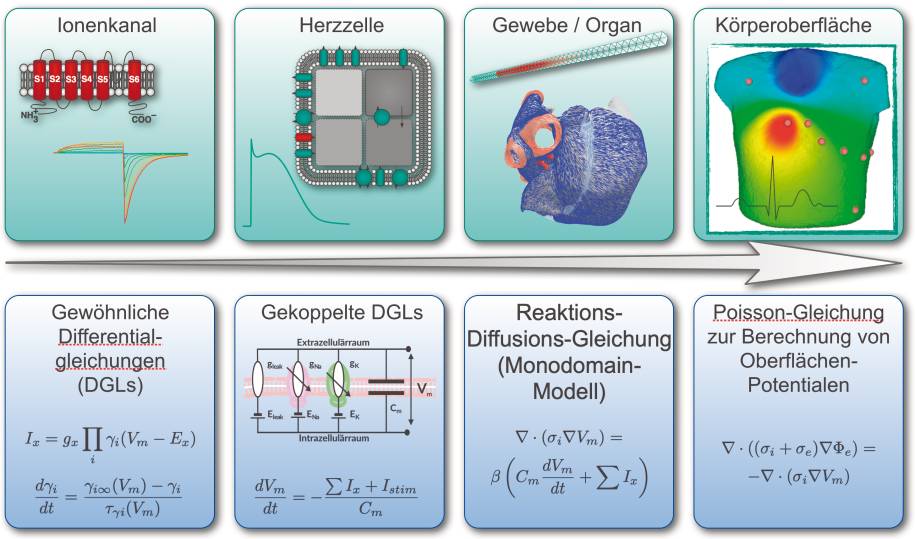
Die Elektrophysiologiedes Herzens wird in vier Stufen simuliert: Ionen, Zelle,Gewebe, Körper(Bild 1)
Autor
Stufe 1: Ionen
Die Träger der elektrischen Energie im Herzen sind Ionen, hauptsächlich Calcium, Kalium und Natrium. Im Herzen löst der Sinusknoten den elektrischen Impuls aus. Dieser Impuls überträgt sich über die Herzzellen und verursacht die Pumpbewegung des Herzens.Irgendwie müssen die Ionen also durch die Herzzellen hindurchwandern können, sonst wäre es nicht möglich, dass der elektrische Impuls sich über das Herz ausbreitet. Die elektrische Kommunikation zwischen Herzzellen funktioniert über die Zellmembran. In der Zellmembran sind Ionenkanäle in der Größenordnung von einigen Nanometern eingelassen. Ionen können über solche Ionenkanäle aus einer Zelle heraus in die nächste Zelle hinein.Auf diese Weise findet der Transport von elektrischer Energie statt. In einer Herzzelle gibt es circa 20 Typen von Ionenkanälen für unterschiedliche Arten von Ionen. In Bild 1 sehen Sie links oben die schematische Darstellung eines Ionenkanals. In der zweiten Spalte mit der Herzzelle stehen die länglichen grünen Symbole für die Ionenkanäle in der Zellmembran. Wie diese Vorgänge innerhalb der Zeit ablaufen, lässt sich mithilfe von Differentialgleichungen beschreiben. Auf diese Weise gelingt ihre Simulation.Stufe 2: Die einzelne Herzzelle
Eine Zelle enthält zahlreiche Ionenkanäle. Neben den passiv arbeitenden Ionenkanälen sind außerdem auch die aktiv wirkenden Ionenpumpen zu berücksichtigen. Die Herzsimulation simuliert nicht jeden Ionenkanal jeder Zelle einzeln, sondern fasst den Austausch, der über die Ionenkanäle einer Zelle läuft, zu einem Wert zusammen.Das ist möglich, denn von der Elektrophysiologie her handelt es sich um eine Parallelschaltung der Ionenkanäle. Die mathematische Simulation ist durch gekoppelte Differentialgleichungen möglich. In Bild 1 sieht man im unteren Teil von Spalte B das elektrische Ersatzschaltbild für die Elektrophysiologie einer Zelle.An dieser Stelle drängt sich fast zwangsläufig die Frage auf, wie man auf so etwas kommt. Nun, dafür bedarf es der Forschungen gleich mehrerer Nobelpreisträger. Die grundlegenden Forschungsarbeiten zur Elektrophysiologie stammen aus dem Jahr 1952 und wurden von A. L. Hodgkin und A. F. Huxley durchgeführt (vergleiche Kasten Der lange Weg zum Herzsimulator).Für ihre Untersuchungen verwendeten die Forscher spezielle Nervenzellen von Tintenfischen, die sogenannten Riesenaxone, die bis zu eintausendmal dicker werden können als die Zellen von Säugetieren. Die schiere Größe dieser Zellen ermöglichte es den Wissenschaftlern, an ihnen elektrische Messungen durchzuführen.Der lange Weg zum Herzsimulator
<b>1843 </b>
Den Forschern gelang es, in einem mathematischen Modell zu beschreiben, wie das Öffnen und Schließen der Ionenkanäle zusammenhängt mit der elektrischen Spannung, dem Membranwiderstand und dem daraus resultierenden Strom. Das ist das sogenannte Hodgkin-Huxley-Modell [2]. 1963 haben die Forscher für die Entwicklung dieses Modells den Medizin-Nobelpreis erhalten.Noch eine Stufe tiefer dringt die „Patch-Clamp-Technik“ [3] vor. Damit lässt sich sogar der Strom einzelner Ionenkanäle einer Zellmembran messen. Diese Technik wurde erstmals 1976 von Erwin Neher und Bert Sakmann beschrieben, die dafür 1991 den Medizin-Nobelpreis bekamen. Die Herzsimulation am KIT basiert grundlegend auf diesen Forschungsarbeiten. Ohne sie wäre ein Herzsimulator undenkbar.Bereits auf der Ebene einzelner Zellen ergeben sich gute Anwendungsmöglichkeiten mit klinischer Relevanz. Medikamente können beispielsweise das Öffnen und Schließen bestimmter Ionenkanäle beeinflussen.Wenn man ihre Wirkung im Modell simulieren will, geht man folgendermaßen vor: Zunächst benötigt man Messergebnisse. Dafür bringen die Forscher an einer im Labor isolierten Zelle das Medikament an und messen mit der Patch-Clamp-Technik die elektrischen Auswirkungen auf die Ionenkanäle. Die Messergebnisse übertragen sie dann in das Modell, indem sie die entsprechenden Differentialgleichungen anpassen.Sobald das Modell die gemessenen Werte korrekt reproduzieren kann, lassen die Forscher die komplette Herzsimulation ablaufen. Auf diese Weise können sie erkennen, welche Auswirkungen eine Änderung auf molekularer Ebene auf die Herztätigkeit insgesamt haben würde.
Ebene 3: Das Gewebe des Organs
Auf der nächsthöheren Stufe werden Herzzellen zum Gewebe zusammengefasst. Zusätzlich zum Zellgewebe ist als weiterer relevanter Parameter die Dicke der Herzwand zu berücksichtigen. Auf dieser Stufe hat man es mit einer dreidimensionalen Simulation zu tun, die mithilfe einer Finite-Elemente-Methode gelingt. Die Herzsimulation fasst jeweils circa 1000 biologische Zellen zu einem „finiten Element“ zusammen. Für eine realistische Herzsimulation werden zwischen 2 Millionen und 10 Millionen solcher Elemente benötigt.Für die Berechnung, wie sich die Erregungswelle über das Herz ausbreitet, verwenden die Karlsruher Forscher eine Reaktions-Diffusions-Gleichung. Die Erregung diffundiert aus einer Zelle zur Nachbarzelle, die ihrerseits reagiert. Im Prinzip funktioniert diese Ausbreitung wie eine La-Ola-Welle im Fußballstadion.Ebene 4: Einbettung des Organs in den Körper
Im vierten Schritte bettet man das schlagende Herz in den Körper ein. Berechnet wird, an welchen Stellen der Körperoberfläche welches elektrische Oberflächenpotenzial resultiert. Für die Berechnung kommen Poisson-Gleichungen zum Einsatz. Auf diese Weise lässt sich ein simuliertes EKG ermitteln, das sich anschließend gut mit dem real gemessenen EKG vergleichen lässt. Auf diese Weise gelingt es, die Vorgänge auf molekularer Ebene mit den Messergebnissen eines EKG zu verbinden. Wenn ein Gendefekt beispielsweise die Arbeitsweise eines Ionenkanals verändert, dann kann die Herzsimulation das daraus resultierende EKG vorhersagen.Vom einzelnen Ion bis zum EKG
Was an der Herzsimulation faszinieren kann, ist die Durchlässigkeit der Simulation vom einzelnen Ion im molekularen Bereich bis hin zum EKG, das der Arzt an der Körperoberfläche des Patienten misst. Man könnte einwenden: Es kann nicht anders sein. Denn irgendwo muss die elektrische Spannung, die sich im EKG messen lässt, schließlich herkommen. Natürlich stimmt der Einwand. Aber beeindruckend ist diese Durchlässigkeit dann doch.Modellierung der räumlichen Gestalt
Bis hierhin ging es um die Modellierung der Elektrophysiologie. Ergänzend zu den elektrischen Vorgängen simulieren die Forscher auch die biomechanischen Vorgänge. Die elektrisch aktivierten Zellen verformen sich und bilden in ihrer Gesamtheit eine Pumpe, die das Blut durch den Kreislauf transportiert. Bild 2 zeigt einen Ausschnitt der biomechanischen Simulation.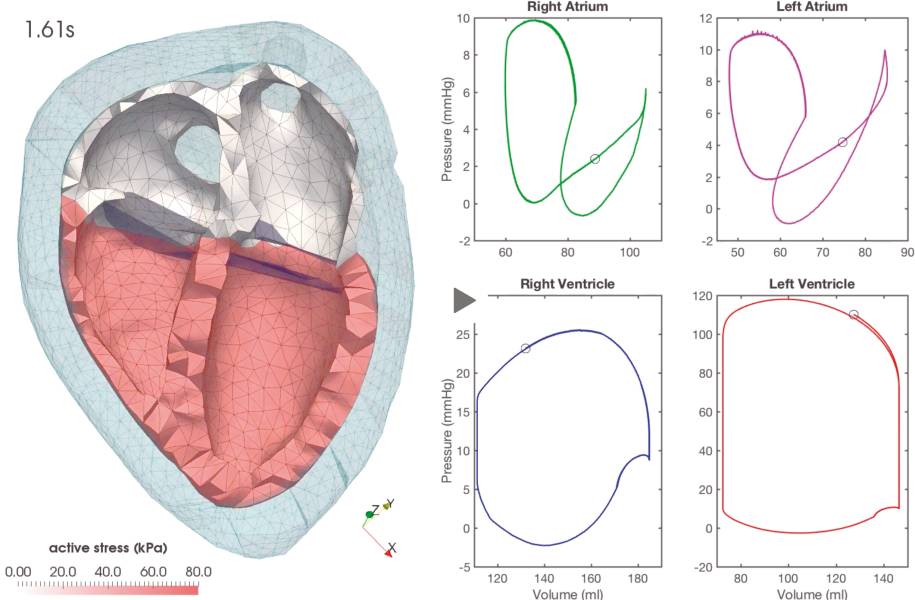
Biomechanische Simulationder vier Herzkammern. Verlauf von Volumen und Druck innerhalb der Kammern(Bild 2)
Autor
Im linken Teil der Abbildung sehen Sie die räumliche Modellierung der beiden Vorhöfe und der beiden Herzkammern. Dazu entsprechend zeigt rechts jeweils ein Schema den Verlauf von Volumen und Druck innerhalb einer Kammer im zeitlichen Verlauf eines Herzschlags.Der Ablauf sei am Beispiel der linken Herzkammer (Left Ventricle) kurz erklärt. Die Aufgabe der linken Herzkammer ist es, frisches, sauerstoffreiches Blut in den Blutkreislauf zu pumpen.Die rote Linie zeigt ausgehend vom unteren linken Punkt folgenden Verlauf:
- Zunächst liegen die Werte sowohl für den Druck als auch das Volumen nahe bei null.
- Das Volumen der Herzkammer steigt dann an, bis die Kammer ihr maximales Fassungsvermögen erreicht hat. Währenddessen ändert sich der Druck nur geringfügig.
- Es fließt weiter Blut in die Kammer, die Kammer kann sich aber nicht weiter ausdehnen, also erhöht sich der Druck.
- Bei einem bestimmten Druck öffnet sich die Herzklappe und Blut kann abfließen.
- Zunächst bleibt der Druck noch einigermaßen gleich, aber das Volumen verringert sich wieder, da das Blut in den Körperkreislauf gepumpt wird.
- Dann verringert sich auch der Druck, bis auch er wieder am unteren Ausgangspunkt angekommen ist.
Implementierung
Das ist also das Modell des Herzens. Und nun geht es an die Implementierung des Modells in Form von Software. Das Team hat das Modell in C++ umgesetzt. Entsprechend dem Aufbau der Modellierung zerfällt auch die Implementierung in mehrere Teile, siehe dazu Tabelle 1.Tabelle 1: Implementierung der Herzsimulation
|
Die Tabelle zeigt für vier Ebenen die mathematische Umsetzung, die eingesetzten Softwarebibliotheken sowie die Größenordnung des daraus resultierenden Rechenaufwands. Auf der Ebene von Ionenkanal und Zelle wird die Elektrophysiologie simuliert. Auf der Ebene von Gewebe und Körper wird darüber hinaus die biomechanische Situation simuliert.Die Simulation des Gewebes stellte das Team vor besondere Herausforderungen. Für Finite-Elemente-Berechnungen sind zwar fertige Lösungen auf dem Markt erhältlich, aufgrund der Besonderheiten der Anwendung waren die Forscher allerdings mit keiner der verfügbaren Lösungen zufrieden. Daher entschlossen sie sich, eine eigene, maßgeschneiderte Lösung zu entwickeln.Die Simulation des Gewebes stellt den mit Abstand rechenaufwendigsten Teil der gesamten Simulation dar. Während die anderen Teile der Simulation in Sekundenbruchteilen erledigt sind, erfordern die Finite-Elemente-Berechnungen mehrere Stunden. Für die Simulation eines Herzschlags, der ungefähr eine Sekunde dauert, berechnet die Anwendung 100 000 Teilschritte zu je 10 µs Länge. Eine Ausnahme bilden die Poisson-Gleichungen zur Berechnung des EKG. Diese werden nur einmal pro Millisekunde gelöst, weil dies der Auflösung eines EKG entspricht.
Abstrakte Basisklasse
Und wie sind Zellen implementiert? Siehe da, es ist tatsächlich so, wie in der Tagtraumfantasie vorgestellt: Für Zellen gibt es eine abstrakte Basisklasse, die Listing 1 zeigt. Von dieser abstrakten Basisklasse werden anschließend unterschiedliche Zelltypen abgeleitet.Listing 1: Abstrakte Basisklasse für eine Herzzelle
template<class T>
class vbElphyModel
{
public:
vbElphyModel(void) {};
virtual ~vbElphyModel(void) {};
virtual void Init(void) = 0;
virtual void Print(ostream &tempstr, double t, T V)
= 0;
virtual void LongPrint(ostream &tempstr, double t,
T V) = 0;
virtual void GetParameterNames(vector<string> &)
= 0;
virtual void GetLongParameterNames(vector<string> &)
= 0;
virtual T GetVm(void) = 0;
virtual T GetCai(void) = 0;
virtual T GetCao(void) = 0;
virtual T GetNai(void) = 0;
virtual T GetNao(void) = 0;
virtual T GetKi(void) = 0;
virtual T GetKo(void) = 0;
virtual int GetSize(void) = 0;
virtual T* GetBase(void) = 0;
virtual T Volume(void) = 0; // in m^3
virtual T GetAmplitude(void) = 0; // in nA
virtual T GetStimTime(void) = 0; // in s
virtual void alloc_ida() {};
virtual void SetCai(T) {};
virtual void SetCao(T) {};
virtual void SetNai(T) {};
virtual void SetNao(T) {};
virtual void SetKi(T) {};
virtual void SetKo(T) {};
virtual void SetTinc(T) {};
virtual inline bool OutIsTCa() { return false; }
virtual inline T GetTCa() { return 0; }
inline void WriteStatus(FILE *writeto) {
kaWrite(GetBase(), (int) sizeof(ML_CalcType),
(size_t)GetSize() / sizeof(ML_CalcType), writeto);
};
inline void ReadStatus(FILE *readfrom) {
kaRead(GetBase(), (int) sizeof(ML_CalcType),
(size_t)GetSize() / sizeof(ML_CalcType), readfrom);
};
virtual inline void Set(vbElphyModel &from) {
memcpy(GetBase(), from.GetBase(), from.GetSize());
};
virtual int GetNumStatus() { return 0; };
virtual void GetStatus(double *p) const {};
virtual void SetStatus(const double *p) {};
virtual T Calc(double tinc, T V, T i_external
= 0., T stretch = 1., int euler = 1) = 0;
virtual T CalcRungeKutta2(double tinc, T V,
T i_external = 0., T stretch = 1., int euler = 1)
{
double status[255], status2[255], status3[255];
GetStatus(status);
double dV = Calc(tinc*.5, V, i_external, stretch,
euler);
GetStatus(status2);
dV = Calc(tinc, V + dV, i_external, stretch,
euler);
GetStatus(status3);
for (int i = 0; i < GetNumStatus(); i++)
status[i] += (status3[i] - status2[i]);
SetStatus(status);
return dV;
}
virtual T CalcRungeKutta4(double tinc, T V,
T i_external = 0., T stretch = 1., int euler = 1)
{
double status1[255], status2[255], status4[255],
status6[255], status7[255], status8[255];
GetStatus(status1);
double dV1 = Calc(tinc*.5, V, i_external, stretch,
euler);
GetStatus(status2);
double dV2 = Calc(tinc*.5, V + dV1, i_external,
stretch, euler);
GetStatus(status4);
double dV3 = Calc(tinc, V + dV2, i_external,
stretch, euler);
GetStatus(status6);
for (int i = 0; i < GetNumStatus(); i++)
status7[i] = status1[i] + (status6[i] -
status4[i]);
SetStatus(status7);
double dV4 = Calc(tinc, V + dV3, i_external,
stretch, euler);
GetStatus(status8);
for (int i = 0; i < GetNumStatus(); i++)
status1[i] += (status2[i] - status1[i]) / 3.
+ (status4[i] - status2[i]) / 1.5
+ (status6[i] - status4[i]) / 3.
+ (status8[i] - status7[i]) / 6.;
SetStatus(status1);
return dV1 / 3. + dV2 / 1.5 + dV3 / 3. + dV4 / 6.;
}
virtual inline bool AddHeteroValue(string desc,
double val) {
cerr << "AddHeteroValue is not defined in the
corresponding cell model!\n";
return false;
};
};
Bereits ein kurzer Blick in den Code wird viele Fragen aufwerfen, die dieser Artikel nicht beantworten will und kann. Genau hier sieht Dr. Loewe aber auch ein Problem des gesamten Projekts.Vom Lebenszyklus her gesehen hat die Software zum aktuellen Zeitpunkt streckenweise noch den Charakter eines Prototyps. Die Software ist so komplex und umfangreich geworden, dass inzwischen nur noch wenige Mitarbeiter einen Überblick über das gesamte Projekt haben. Das ist generell ein kritischer Moment im Leben einer Software, nicht nur bei diesem Projekt.Damit es nicht dabei bleibt, hat das Team um Dr. Loewe ein eigenes Forschungsprojekt aufgesetzt. Bereits seit 2018 läuft das Projekt „SuLMaSS“, das steht für „Sustainable Lifecycle Management for Scientific Software“. Im Rahmen dieses von der Deutschen Forschungsgemeinschaft (DFG) geförderten Projekts entwickelt man am KIT gemeinsam mit den Universitäten Freiburg, Graz und Bordeaux die Infrastruktur für ein nachhaltiges Lifecycle-Management von Forschungssoftware. Auf diese Weise werden gleich zwei Fliegen mit einer Klappe geschlagen:
- Die wissenschaftliche Community erhält ein allgemein verwendbares Lebenszyklusmodell.
- Als Proof of Concept für dieses Modell wird die Software für Herzsimulation entsprechend diesem Konzept konsolidiert.
Beispiel Herzrhythmusstörungen
Damit ist das Herz abstrakt modelliert und in Software implementiert. Aber ist diese Simulation auch in der klinischen Praxis zu etwas nütze? In einem medizinischen Fachartikel haben die Forscher ein Anwendungsbeispiel vorgestellt [8]. Es geht es um die Therapie von Herzrhythmusstörungen.Es gibt unterschiedliche Arten von Herzrhythmusstörungen. Am weitesten verbreitet ist das Vorhofflimmern. In Deutschland leiden daran circa 300 000 Menschen, in ganz Europa sind es über 6 Millionen. Das Prinzip der Krankheit sei kurz erklärt.Ein gesunder Herzschlag läuft so ab: Im Herzen löst der Sinusknoten einen elektrischen Impuls aus. Dieser breitet sich wellenförmig über das Herz aus und aktiviert auf diese Weise das Muskelgewebe, das dadurch eine Pumpbewegung ausführt. Nachdem der elektrische Impuls durch das Herz gewandert ist, verebbt die Welle und es ist wieder Ruhe im Herzen, bis der nächste Impuls beginnt. Und so geht das ein Leben lang.Beim Vorhofflimmern verebbt der elektrische Impuls jedoch nicht vollständig. Es tritt sozusagen keine Ruhe im Herzen ein, sondern das Muskelgewebe bleibt ständig in einer leichten elektrischen Erregung, es flimmert, wobei sich das Flimmern entlang von individuell verschiedenen Pfaden verbreitet. Patienten mit Vorhofflimmern klagen über unspezifische Beschwerden wie Müdigkeit, Schlafstörungen, Herzrasen. Ihr Schlaganfallrisiko ist erhöht.Herkömmliche Therapien
Eine Art der Behandlung ist die Gabe von Medikamenten. Eine andere Art ist ein Eingriff im Herzen, bei dem die flimmernden Pfade im Herzen mit Hitze, Kälte oder Laser stellenweise verödet werden, das Verfahren wird Ablation genannt. Anschließend können die Flimmerimpulse sich nicht mehr ausbreiten und das Flimmern ist beseitigt. Diese invasiven Eingriffe funktionieren zwar einigermaßen gut, haben aber oft Nebenwirkungen. In vielen Fällen treibt man nämlich den Teufel mit Beelzebub aus, weil die Patienten einige Zeit nach der Operation Vorhofflattern entwickeln, eine andere Art von Herzrhythmusstörung. Genau hier ist die Medizin gerade an einem Punkt, an dem sie nicht so richtig weiter weiß.Bevor der Kardiologe bei einem Patienten eine solche Ablation vornimmt, wüsste er gern, wie hoch bei dem konkreten Patienten die Wahrscheinlichkeit ist, dass er anschließend Vorhofflattern entwickelt. Bei einem entsprechend hohen Risiko würde er die Operation unterlassen. Bislang kann man dieses Risiko jedoch nicht einschätzen.Erst mal gucken
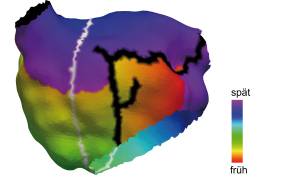
Und genau hier treten die Karlsruher Forscher auf den Plan und schlagen eine Simulation vor. Die Herzsimulation ist mittlerweile extrem präzise und lässt sich darüber hinaus auf die individuellen Gegebenheiten eines bestimmten Patienten anpassen. Man kann beispielsweise die anatomische Form nachbilden und auch die Gabe von Medikamenten berücksichtigen.In der Simulation können die Forscher an einzelnen Punkten in einem Herzvorhof eine Ablation durchführen und die Auswirkungen beobachten. Möglicherweise bilden sich neue Pfade, an denen Vorhofflattern entsteht.Bild 3 zeigt in der Simulation einen solchen Pfad. Wenn kein Flatterpfad entsteht, dann konnten die Forscher damit diejenigen Punkte bestimmen, an denen eine Ablation vorgenommen werden sollte. So lässt sich das Risiko minimieren, dass durch die Bekämpfung des Vorhofflimmerns anschließend Vorhofflattern entsteht.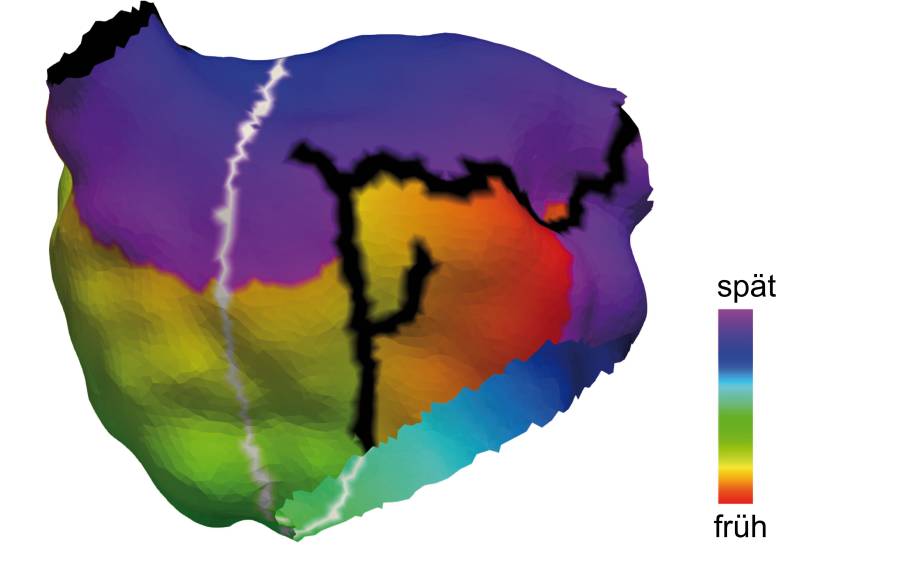
Anatomisches Modelldes linken Vorhofs einer 70-jährigenPatientin. Die Simulation zeigt einen Pfad, an dem sich Vorhofflattern entwickelt(Bild 3)
Autor
Das ist nur ein Beispiel für eine klinisch relevante Anwendung des Herzsimulators. Zahlreiche weitere Anwendungsfälle sind möglich und werden künftig in die klinische Praxis vordringen.
Ausblick
War das nicht ein spannender Ausflug in die Forschung? Und in wenigen Monaten soll der Herzsimulator sogar als Open-Source-Software zur Verfügung stehen!Dann kann die dotnetpro im Rahmen ihres Programmier-dojos die Aufgabe stellen: Und heute messen wir einmal unser eigenes EKG und zeigen passend dazu unser Herz im Simulator an.Fussnoten
- Karlsruher Institut für Technologie (KIT), Institut für Biomedizinische Technik, https://www.ibt.kit.edu/
- Hodgkin-Huxley-Modell, https://de.wikipedia.org/wiki/Hodgkin-Huxley-Modell
- Patch-Clamp-Technik, https://de.wikipedia.org/wiki/Patch-Clamp-Technik
- KIT, Institut für Biomedizinische Technik, Computermodell des Herzens, https://www.ibt.kit.edu/camo.php
- Numerik-Bibliothek CVODE, https://computing.llnl.gov/projects/sundials/cvode
- Numerik-Bibliothek PETSc, Portable, Extensible Toolkit for Scientific Computation,
- Cardiac Electrophysiology Simulator, Verfügbarkeit für März 2020 angekündigt, https://opencarp.org
- Axel Loewe et al., Patient-Specific Identification of Atrial Flutter Vulnerability – A Computational Approach to Reveal Latent Reentry Pathways, Frontiers in Physiology, 14. Januar 2019, https://www.frontiersin.org/journals/physiology/articles/10.3389/fphys.2018.01910/full